CREB
CREB(cAMP response element binding protein)[1]は転写因子であり、cAMP応答配列(cAMP response element、CRE)と呼ばれる特定のDNA配列に結合し、遺伝子の転写を増加させたり低下させたりする[2]。CREBは1987年に、ソマトスタチン遺伝子を調節するcAMP応答性転写因子として最初に記載された[3]。
CREBによって転写が調節される遺伝子には、c-Fos、BDNF、チロシンヒドロキシラーゼ、多くの神経ペプチド(ソマトスタチン、エンケファリン、VGF、副腎皮質刺激ホルモン放出ホルモン)の遺伝子や[2]、哺乳類の概日時計に関与する遺伝子(PER1、PER2)などがある[4]。
CREBの構造と機能は、CREMやATF1などのタンパク質と密接に関連している。CREBタンパク質はヒトを含む多くの動物で発現している。
CREBは脳の神経可塑性や長期記憶の形成に関する役割がよく知られており、空間記憶の形成に不可欠であることが示されている[5]。CREBのダウンレギュレーションはアルツハイマー病の病理への関与が示唆されており、CREBの発現の増加はアルツハイマー病の治療標的の可能性の1つとして考えられている[6]。CREBは哺乳類の光同調にも関与している。
サブタイプ
編集CREBまたはCREB様タンパク質には次のようなものがある
構造
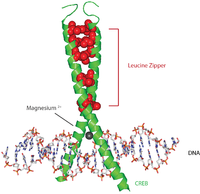
編集CREBタンパク質は、PKA、CAMKなどさまざまなキナーゼによってセリン残基がリン酸化されることで活性化される[7]。CREBタンパク質は活性化されると、他の転写コアクチベーターをリクルートして5' UTRのCREプロモーターに結合する。CREBタンパク質のαヘリックスには疎水的なロイシン残基が並んでいる。これらのロイシン残基は二量体を形成している他方のCREBタンパク質のロイシン残基と強固に結合する。このロイシン残基の連鎖によってロイシンジッパーモチーフが形成される。また、CFREBタンパク質はDNAへの結合を促進するマグネシウムイオンを結合する。
cAMP応答配列
編集cAMP応答配列(CRE)はCREBが結合する応答エレメントであり、高度に保存されたヌクレオチド配列5'-TGACGTCA-3'を含んでいる。CREは通常、遺伝子の上流のプロモーター領域やエンハンサー領域に位置する[8]。ヒトゲノム上には、CREBの結合のhalf-siteとなりうる回文型CREは約750,000個存在する。しかし、これらの部位の大部分はタンパク質の結合を物理的に阻害するシトシンのメチル化のため、非結合状態となっている[9]。
作用機序
編集CREBの典型的な(ただし多少単純化された)作用機序は次のようなものである。シグナルが細胞表面に到達すると対応する受容体が活性化され、cAMPやCa2+などのセカンドメッセンジャーが産生されてプロテインキナーゼが活性化される。プロテインキナーゼは細胞核へ移行し、そこでCREBタンパク質を活性化する。活性化されたCREBタンパク質はCRE領域に結合し、その後コアクチベーターであるCREB結合タンパク質(CBP)が結合して特定の遺伝子をオンにしたりオフにしたりする。CREBのDNAへの結合はbZIPドメインを介して行われる。
脳における機能
編集CREBはさまざまな器官で多くの機能を持っているが、その機能の一部は脳と関係した研究が行われている[10]。神経細胞内のCREBタンパク質は、長期記憶の形成に関与していると考えられており[11]、このことはアメフラシ、キイロショウジョウバエ、ラット、マウスで示されている[1](認知機能におけるCREBを参照)。CREBは、長期増強の後期に必要である。また、CREBは薬物依存症の発症やさらには精神的依存にも重要な役割を果たしている[12][13][14]。
疾患との関係
編集脳でのCREBの阻害はハンチントン病の発症と進行に寄与する場合がある。
CREBのKIDドメインと相互作用するCREB結合タンパク質(CBP)の異常はルビンシュタイン・テイビ症候群と関係している。
CREBの機能低下が大うつ病性障害と関係していることを示唆する証拠がいくつかある[15]。歯状回でCREBを過剰発現させたうつ病のラットは、抗うつ薬を投与したラットと同様の行動をとった[16]。また、未治療の大うつ病性障害患者の大脳皮質では、健常人や抗うつ薬治療を受けた患者と比較して、CREBの濃度が低下していることが剖検から示されている[16]。CREBの機能は、セロトニンやノルアドレナリンがシナプス後のGタンパク質共役受容体に結合することで生じるシグナル伝達経路を介して調節される。これらの神経伝達物質の機能異常も大うつ病性障害への関与が示唆されている[15]。
概日リズムへの関与
編集哺乳類の概日時計の同調は、光によるPERの誘導によって確立される。光はメラノプシンを含む内因性光感受性網膜神経節細胞を興奮させ、網膜視床下部路(RHT)を介して視交叉上核(SCN)へシグナルを伝達する。RHTの興奮によってグルタミン酸が放出され、SCNのNMDA受容体に受容されることで、SCNへのカルシウムの流入が引き起こされる。カルシウムはCaMKの活性を誘導し、PKA、PKC、CK2の活性化が引き起こされる[17]。これらのキナーゼはCREBを概日的にリン酸化し、下流の遺伝子発現をさらに調節する[18]。リン酸化されたCREBはCREを認識し、哺乳類の概日時計を制御する2つの遺伝子、Per1とPer2の転写因子として機能する。このPERタンパク質の誘導によって明暗サイクルに合わせて概日時計を同調させることができ、転写-翻訳フィードバックループを介して自身の転写を抑制して概日時計を進めたり遅らせたりすることができる。しかし、PER1およびPER2タンパク質の誘導の応答性は主観的夜間にのみ顕著である[4]。
CREBの概日リズムへの関与の発見
編集Michael Greenbergは、1993年に光パルスとCREBのリン酸化の相関を示す一連の実験を通してCREBが哺乳類の概日時計に果たす役割を初めて実証した。In vitroでは、主観的夜間の光はCREBのタンパク質レベルではなくリン酸化の増加をもたらした。In vivoでは、主観的夜間に概日リズムの位相シフトを誘導する光パルスはSCNでのCREBのリン酸化と相関していた[19]。2002年のGunther Schutzによる実験では、リン酸化部位であるSer142を欠く変異マウスでは、光パルスに反応した時計調節遺伝子mPer1の誘導を行うことができなかった。さらに、これらの変異マウスは明暗サイクルに同調することが困難であった[20]。
出典
編集- ^ a b Bourtchuladze (1994). “Deficient long-term memory in mice with a targeted mutation of the cAMP-responsive element-binding protein”. Cell 79 (1): 59–68. doi:10.1016/0092-8674(94)90400-6. PMID 7923378.
- ^ a b Purves, Dale; George J. Augustine; David Fitzpatrick; William C. Hall; Anthony-Samuel LaMantia; James O. McNamara & Leonard E. White (2008). Neuroscience (4th ed.). Sinauer Associates. pp. 170–6. ISBN 978-0-87893-697-7
- ^ Montminy, MR; Bilezikjian, LM (1987). “Binding of a nuclear protein to the cyclic-AMP response element of the somatostatin gene.”. Nature 328 (6126): 175–178. Bibcode: 1987Natur.328..175M. doi:10.1038/328175a0. PMID 2885756.
- ^ a b Dibner, Charna; Schibler, Ueli; Albrecht, Urs (2010). “The Mammalian Circadian Timing System: Organization and Coordination of Central and Peripheral Clocks”. Annual Review of Physiology 72 (1): 517–549. doi:10.1146/annurev-physiol-021909-135821. PMID 20148687.
- ^ Silva, A. J.; Kogan, J. H.; Frankland, P. W.; Kida, S. (1998). “CREB and memory”. Annual Review of Neuroscience 21: 127–148. doi:10.1146/annurev.neuro.21.1.127. ISSN 0147-006X. PMID 9530494.
- ^ Pugazhenthi, Subbiah; Wang, Maorong; Pham, Serena; Sze, Chun-I.; Eckman, Christopher B. (2011-08-19). “Downregulation of CREB expression in Alzheimer's brain and in Aβ-treated rat hippocampal neurons”. Molecular Neurodegeneration 6: 60. doi:10.1186/1750-1326-6-60. ISSN 1750-1326. PMC 3174124. PMID 21854604.
- ^ Shaywitz, Adam J.; Greenberg, Michael E. (1999). “CREB: A Stimulus-Induced Transcription Factor Activated by A Diverse Array of Extracellular Signals”. Annual Review of Biochemistry 68 (1): 821–861. doi:10.1146/annurev.biochem.68.1.821. PMID 10872467.
- ^ Carlezon, WA; Duman, RS; Nestler, EJ (August 2005). “The many faces of CREB”. Trends in Neurosciences 28 (8): 436–445. doi:10.1016/j.tins.2005.06.005. PMID 15982754.
- ^ Altarejos, Judith Y.; Montminy, Marc (March 2011). “CREB and the CRTC co-activators: sensors for hormonal and metabolic signals”. Nature Reviews Molecular Cell Biology 12 (3): 141–151. doi:10.1038/nrm3072. ISSN 1471-0072. PMC 4324555. PMID 21346730.
- ^ “The many faces of CREB”. Trends in Neurosciences 28 (8): 436–45. (August 2005). doi:10.1016/j.tins.2005.06.005. PMID 15982754.
- ^ Kandel, Eric R. (2012-05-14). “The molecular biology of memory: cAMP, PKA, CRE, CREB-1, CREB-2, and CPEB”. Molecular Brain 5: 14. doi:10.1186/1756-6606-5-14. ISSN 1756-6606. PMC 3514210. PMID 22583753.
- ^ “Sex differences in basal and cocaine-induced alterations in PKA and CREB proteins in the nucleus accumbens”. Psychopharmacology 203 (3): 641–50. (April 2009). doi:10.1007/s00213-008-1411-5. PMID 19052730.
- ^ “CREB regulation of BK channel gene expression underlies rapid drug tolerance”. Genes, Brain, and Behavior 8 (4): 369–76. (June 2009). doi:10.1111/j.1601-183X.2009.00479.x. PMC 2796570. PMID 19243452.
- ^ “A role for calmodulin-stimulated adenylyl cyclases in cocaine sensitization”. Journal of Neuroscience 29 (8): 2393–403. (February 2009). doi:10.1523/JNEUROSCI.4356-08.2009. PMC 2678191. PMID 19244515.
- ^ a b Belmaker, R. H.; Agam, Galila (2008). “Major depressive disorder”. New England Journal of Medicine 358 (1): 55–68. doi:10.1056/nejmra073096. PMID 18172175.
- ^ a b Blendy, JA (2006). “The role of CREB in depression and antidepressant treatment”. Biol Psychiatry 59 (12): 1144–50. doi:10.1016/j.biopsych.2005.11.003. PMID 16457782.
- ^ Iyer, Rajashekar; Wang, Tongfei; Gillette, Martha (September 19, 2014). “Circadian gating of neuronal functionality: a basis for iterative metaplasticity”. Frontiers in Systems Neuroscience 8: 164. doi:10.3389/fnsys.2014.00164. PMC 4168688. PMID 25285070.
- ^ Obrietan, Karl; Impey, Soren; Smith, Dave; Athos, Jaime; Storm, Derrick R. (April 11, 2002). “Circadian regulation of cAMP response element-mediated gene expression in the suprachiasmatic nuclei.”. Neuron 274 (25): 17748–17756. doi:10.1074/jbc.274.25.17748. PMID 10364217.
- ^ Ginty, D. D.; Kornhauser, J. M.; Thompson, M. A.; Bading, H.; Mayo, K. E.; Takahashi, J. S.; Greenberg, M. E. (April 9, 1993). “Regulation of CREB phosphorylation in the suprachiasmatic nucleus by light and a circadian clock”. Science 260 (5105): 238–241. Bibcode: 1993Sci...260..238G. doi:10.1126/science.8097062. ISSN 0036-8075. PMID 8097062.
- ^ Gau, Daniel; Lemberger, Thomas; von Gall, Charlotte; Kretz, Oliver; Le Minh, Nguyet; Gass, Peter; Schmid, Wolfgang; Schibler, Ueli et al. (April 11, 2002). “Phosphorylation of CREB Ser142 Regulates Light-Induced Phase Shifts of the Circadian Clock”. Neuron 34 (2): 245–253. doi:10.1016/S0896-6273(02)00656-6. PMID 11970866.
関連文献
編集- Lauren Slater (2005). Opening Skinner's Box: Great Psychological Experiments of the Twentieth Century. New York: W. W. Norton & Company. ISBN 978-0-393-32655-0
- “Common molecular mechanisms in explicit and implicit memory”. J. Neurochem. 97 (6): 1520–33. (2006). doi:10.1111/j.1471-4159.2006.03870.x. PMID 16805766.
- “CREB: the unindicted cancer co-conspirator”. Trends Cell Biol. 15 (9): 457–9. (2005). doi:10.1016/j.tcb.2005.07.007. PMID 16084096.
- “Disruption of CREB function in brain leads to neurodegeneration”. Nat. Genet. 31 (1): 47–54. (2002). doi:10.1038/ng882. PMID 11967539.
- “Transcriptional regulation by the phosphorylation-dependent factor CREB”. Nat. Rev. Mol. Cell Biol. 2 (8): 599–609. (2001). doi:10.1038/35085068. PMID 11483993.
- “CREB as a memory modulator: induced expression of a dCREB2 activator isoform enhances long-term memory in Drosophila”. Cell 81 (1): 107–15. (1995). doi:10.1016/0092-8674(95)90375-5. PMID 7720066.
- “Induction of a dominant negative CREB transgene specifically blocks long-term memory in Drosophila”. Cell 79 (1): 49–58. (1994). doi:10.1016/0092-8674(94)90399-9. PMID 7923376.
外部リンク
編集- http://www.ebi.ac.uk/interpro/entry/IPR001630
- Johannessen, M., Pedersen Delghandi, M., and Moens, U. (2004) - What Turns CREB on ? - Cell Signall.; 10:1211-1227. https://web.archive.org/web/20070928090058/http://www.sigtrans.org/publications/what-turns-creb-on/
- https://web.archive.org/web/20060902183214/http://focus.hms.harvard.edu//2001/Oct26_2001/neuroscience.html
- CREB Protein - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス(英語)
- Drosophila Cyclic-AMP response element binding protein A - The Interactive Fly
- Drosophila Cyclic-AMP response element binding protein B at 17A - The Interactive Fly