5SリボソームRNA
5SリボソームRNA(英: 5S ribosomal RNA、略称: 5S rRNA)は、約120ヌクレオチドからなるリボソームRNA分子で、約 40 kDaである。生命の全ドメイン(細菌、古細菌、真核生物)のリボソームの大サブユニットの構造的・機能的構成要素であるが、菌類と動物のミトコンドリアのリボソームには存在しない。「5S」は超遠心分析における分子の沈降係数を意味しており、スヴェドベリ(S)を単位として表したものである[1]。
| 5SリボソームRNA | |
|---|---|
 | |
| 識別 | |
| 略称 | 5S_rRNA |
| Rfam | RF00001 CL00113 |
| その他のデータ | |
| リボ核酸の種類 | 遺伝子; rRNA |
| ドメイン | 真核生物; 細菌; 古細菌 |
| GO | 0005840 0003735 |
| SO | 0000652 |
| PDB構造 | PDBe |

生合成
編集原核生物では、5S rRNAの遺伝子は典型的にはrRNAオペロン中、小サブユニットと大サブユニットのrRNA遺伝子の下流に位置し、ポリシストロンの前駆体として共に転写される[3]。真核生物の核ゲノムでは複数の5S rRNA遺伝子コピー(5S rDNA)がタンデムリピートとして密集して存在しており、そのコピー数は種によって異なる[4][5]。真核生物の5S rRNAはRNAポリメラーゼIIIによって合成されるが、他のrRNAはRNAポリメラーゼIによって転写された45S前駆体からの切断によって形成される。ツメガエルXenopusの卵母細胞では、9つのジンクフィンガーからなる転写因子TFIIIAの4番目から7番目のフィンガーが5S rRNAの中心領域に結合する[6][7]。5S rRNAとTFIIIAの結合は、5S rRNAの遺伝子の転写を抑制し、リボソームの組み立てに必要となるまで5S rRNA転写産物を安定化する役割がある[8]。
構造
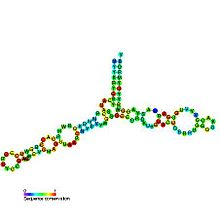
編集5S rRNAの二次構造は5つのヘリックス(I–V)、4つのループ(B–E)、1つのヒンジ(A)からなり、これらがY字状の構造を形成する。ループCとDはヘアピンループ、BとEはインターナルループである[4]。系統学的な研究によると、ヘリックスIとIIIが歴史が古い可能性が高い[9]。ヘリックスIIIには高度に保存された2つのアデノシンが存在する[10]。ヘリックスVはヘアピン構造であり、TFIIIAと相互作用すると考えられている[4]。
リボソーム内での位置
編集免疫電顕法、化学的分子間架橋、X線結晶構造解析などさまざまな分子的技術を用いて、リボソーム大サブユニット(LSU)内の5S rRNAの位置はきわめて正確に決定されている。細菌と古細菌では、LSUは2つのRNA要素(5S rRNAと23S rRNA)と多数の結合タンパク質によって構成されている[3]。真核生物のLSUは5S、5.8S、28S rRNAと、さらに多くのタンパク質が含まれる[12][13]。LSUの立体構造には、 L1 protuberance、central protuberance(CP)、L7/L12 stalkと呼ばれる3つの突出部が存在している。5S rRNAはCPに位置し、この突出部の形成と構造に加わっている。CPの他の主要な構成要素には、23S rRNA(真核生物では28S)とL5、L18、L25、L27などのいくつかのタンパク質がある[14]。
リボソームでの機能
編集5S rRNAの正確な機能はまだ明らかではない。大腸菌Escherichia coliでは、5S rRNAの遺伝子の欠失はタンパク質合成速度を低下させ、他のrRNA遺伝子(16Sや23S)の同程度のコピー数の欠失よりも細胞の適応に関して大きな悪影響を与える[15]。結晶学的な研究により、5S rRNA結合タンパク質やCPに位置する他のタンパク質がtRNAの結合に重要な役割を果たすことが示されている[14]。また、5S rRNAと23 rRNAが位置的、物理的に近接することでリボソームのぺプチジル基転移中心やGTPアーゼ結合中心が形成されており、5S rRNAは、5S rRNA結合タンパク質やCP、大小サブユニット間のブリッジ、tRNA結合部位の他の構成要素とともに、リボソームの2つの機能的中心間の媒介因子として機能していることが示唆されている[14]。
リボソームの組み立てにおける役割
編集真核生物では、細胞質のリボソームは4つのrRNAと80以上のタンパク質から組み立てられる[13][16]。5S rRNAはいったん転写されると、3'末端がエキソヌクレアーゼRex1p、Rex2p、Rex3pによってプロセシングされる[4]。60Sと40Sのサブユニットは核から細胞質へ輸送され、そこで結合して成熟した翻訳活性を持つ80Sリボソームが形成される。5S rRNAがリボソームに組み込まれる正確な時期については議論があるが[4]、5S rRNAは、リボソームタンパク質L5とのリボヌクレオタンパク質複合体として、60S粒子の前駆体である90S粒子へ組み込まれることは一般的に受け入れられている[16]。
タンパク質との相互作用
編集5S rRNAと相互作用する重要なタンパク質のいくつかを下に挙げる。
Laタンパク質
編集5S rRNAとLaタンパク質との相互作用は、RNAを細胞内のエキソヌクレアーゼによる分解から防ぐ[17]。Laタンパク質は全ての真核生物の核内に存在し、RNAポリメラーゼIIIによって転写されるいくつかのタイプのRNAと結合する。Laタンパク質は、これらのRNA(5S rRNAを含む)の3'末端のオリゴウリジントラクト(oligo-uridine tract)を介して結合し、RNAの安定性とフォールディングを補助する[4][18]。
L5タンパク質
編集真核生物では、リボソームタンパク質L5は5S rRNAと結合して安定化し、細胞質にも核にも存在するプレリボソームリボヌクレオタンパク質粒子(pre-ribosomal RNP)を形成する。L5の欠乏は5S rRNAの核への輸送を防ぎ、リボソームの組み立ては低下する[4]。
他のリボソームタンパク質
編集原核生物の5S rRNAはL5、L18、L25リボソームタンパク質と結合するが、真核生物の5S rRNAはL5とのみ結合することが知られている[19]。アフリカ睡眠病の病原体となるトリパノソーマTrypanosoma bruceiでは、5S rRNAは2つの近縁なRNA結合タンパク質P34とP37に結合し、これらが欠失することで5S rRNAの全体的なレベルが低下する[4]。
細胞小器官のリボソーム中の存在
編集ミトコンドリアや色素体(共生細菌に由来する細胞小器官)の翻訳装置と関連する細菌の翻訳装置は多くの特徴を共有しているが、一方で顕著な差異も存在する。細胞小器官のゲノムは例外なく小サブユニットと大サブユニットのrRNAをコードしているが、5S rRNAの遺伝子(rrn5)に関してはそうではない。大部分の色素体のゲノムではrrn5は容易に同定される。対照的に、ミトコンドリアのrrn5は当初は植物と一部の原生生物に限定されていると考えられていた[20][21]。配列組成バイアスや構造的な変化に関する情報を組み込んだ特殊な共変化モデルによって、さらに多様な細胞小器官で5S rRNAが同定された[22]。この解析により、5S rRNAの遺伝子は大部分の原生生物のミトコンドリアゲノムだけでなく、特定のアピコプラスト(トキソプラズマToxoplasma gondiiやコクシジウムEimeria tenellaなどの病原性原生動物の非光合成性色素体)のゲノムにも存在することが示された。
ストラメノパイルのミトコンドリアの5S rRNAは、二次構造の多様性が最も大きい[22]。褐藻で見られる置換型のミトコンドリア5S rRNAは最も変わったケースであり、通常分子の5'末端と3'末端によって構成されるヘリックスIは閉じたヘアピン構造に置き換えられ、開いた3方向のジャンクション構造となっている。
現行の証拠からは、わずかなグループ、特に動物、菌類、アルベオラータとユーグレノゾアのミトコンドリアDNAだけが5S rRNAの遺伝子を欠いていることが示唆されている[22]。通常5S rRNAと結合タンパク質によって占められているCP構造は、これらの生物のミトコンドリアではさまざまに再構成されている。菌類のミトコンドリアリボソームでは、5S rRNAは大サブユニットのrRNAの拡張配列によって置き換えられている[23]。ユーグレノゾアのキネトプラスチドでは、CPは進化的に新規のミトコンドリアリボソームタンパク質によって構成されている[24]。動物のミトコンドリアリボソームでは、特定のミトコンドリアtRNAが失われた5S rRNAに置き換わるように共適応が起こっている[25][26]。
出典
編集- ^ “5S Ribosomal RNA Database”. Nucleic Acids Res. 30 (1): 176–178. (January 2002). doi:10.1093/nar/30.1.176. PMC 99124. PMID 11752286.
- ^ “The 3D arrangement of the 23 S and 5 S rRNA in the Escherichia coli 50 S ribosomal subunit based on a cryo-electron microscopic reconstruction at 7.5 A resolution”. J Mol Biol 298 (1): 35–59. (2000). doi:10.1006/jmbi.2000.3635. PMID 10756104.
- ^ a b Kaczanowska, M; Rydén-Aulin, M (September 2007). “Ribosome biogenesis and the translation process in Escherichia coli”. Microbiology and Molecular Biology Reviews 71 (3): 477–494. doi:10.1128/MMBR.00013-07. PMC 2168646. PMID 17804668.
- ^ a b c d e f g h Ciganda, Martin; Williams, Noreen (July 2011). “Eukaryotic 5S rRNA biogenesis”. Wiley Interdisciplinary Reviews: RNA 2 (4): 523–533. doi:10.1002/wrna.74. PMC 3278907. PMID 21957041.
- ^ Douet, J; Tourmente, S (Jul 2007). “Transcription of the 5S rRNA heterochromatic genes is epigenetically controlled in Arabidopsis thaliana and Xenopus laevis”. Heredity 99 (1): 5–13. doi:10.1038/sj.hdy.6800964. PMID 17487217.
- ^ McBryant, SJ; Veldhoen, N; Gedulin, B; Leresche, A; Foster, MP; Wright, PE; Romaniuk, PJ; Gottesfeld, JM (1995). “Interaction of the RNA binding fingers of Xenopus transcription factor IIIA with specific regions of 5 S ribosomal RNA”. Journal of Molecular Biology 248 (1): 44–57. doi:10.1006/jmbi.1995.0201. PMID 7731045.
- ^ Searles, MA; Lu D; Klug A (2000). “The role of the central zinc fingers of transcription factor IIIA in binding to 5 S RNA”. J Mol Biol 301 (1): 47–60. doi:10.1006/jmbi.2000.3946. PMID 10926492.
- ^ Pelham, HRB; Brown DD (1980). “A specific transcription factor that can bind either the 5S RNA gene or 5S RNA”. Proc. Natl. Acad. Sci. USA 77 (7): 4170–4174. doi:10.1073/pnas.77.7.4170. PMC 349792. PMID 7001457.
- ^ Sun, FJ; Caetano-Anollés, G (Nov 2009). “The evolutionary history of the structure of 5S ribosomal RNA”. Journal of Molecular Evolution 69 (5): 430–443. doi:10.1007/s00239-009-9264-z. PMID 19639237.
- ^ DiNitto, JP; Huber, PW (Oct 23, 2001). “A role for aromatic amino acids in the binding of Xenopus ribosomal protein L5 to 5S rRNA”. Biochemistry 40 (42): 12645–12653. doi:10.1021/bi011439m. PMID 11601989.
- ^ “The complete atomic structure of the large ribosomal subunit at 2.4 Å resolution”. Science 289 (5481): 905–920. (2000). doi:10.1126/science.289.5481.905. PMID 10937989.
- ^ Turowski, TW; Tollervey, D (2015). “Cotranscriptional events in eukaryotic ribosome synthesis”. Wiley Interdisciplinary Reviews: RNA 6 (1): 129–139. doi:10.1002/wrna.1263. PMID 25176256.
- ^ a b Yusupova, G; Yusupov, M (February 2014). “High-resolution structure of the eukaryotic 80S ribosome”. Annual Review of Biochemistry 83: 467–486. doi:10.1146/annurev-biochem-060713-035445. PMID 24580643.
- ^ a b c Gongadze, G. M. (7 January 2012). “5S rRNA and ribosome”. Biochemistry (Moscow) 76 (13): 1450–1464. doi:10.1134/S0006297911130062. PMID 22339598.
- ^ Ammons, D; Rampersad, J; Fox, GE (Jan 15, 1999). “5S rRNA gene deletions cause an unexpectedly high fitness loss in Escherichia coli”. Nucleic Acids Research 27 (2): 637–642. doi:10.1093/nar/27.2.637. PMC 148226. PMID 9862991.
- ^ a b Henras, AK; Soudet, J; Gérus, M; Lebaron, S; Caizergues-Ferrer, M; Mougin, A; Henry, Y (Aug 2008). “The post-transcriptional steps of eukaryotic ribosome biogenesis”. Cellular and Molecular Life Sciences 65 (15): 2334–2359. doi:10.1007/s00018-008-8027-0. PMID 18408888.
- ^ Wolin, SL; Cedervall, T (2002). “The La protein”. Annual Review of Biochemistry 71: 375–403. doi:10.1146/annurev.biochem.71.090501.150003. PMID 12045101.
- ^ Maraia, RJ; Intine, RV (2002). “La protein and its associated small nuclear and nucleolar precursor RNAs”. Gene Expression 10 (1–2): 41–57. PMC 5977531. PMID 11868987.
- ^ Moore PB (March 2001). “The ribosome at atomic resolution”. Biochemistry 40 (11): 3243–3250. doi:10.1021/bi0029402. PMID 11258942.
- ^ Bullerwell, CE; Schnare, MN; Gray, MW (March 2003). “Discovery and characterization of Acanthamoeba castellanii mitochondrial 5S rRNA”. RNA 9 (3): 287–292. doi:10.1261/rna.2170803. PMC 1370395. PMID 12592002.
- ^ Bullerwell, CE; Burger, G; Gott, JM; Kourennaia, O; Schnare, MN; Gray, MW (May 2010). “Abundant 5S rRNA-like transcripts encoded by the mitochondrial genome in Amoebozoa”. Eukaryot Cell 9 (5): 762–773. doi:10.1128/EC.00013-10. PMC 2863963. PMID 20304999.
- ^ a b c Valach, M; Burger, G; Gray, MW; Lang, BF (Dec 2014). “Widespread occurrence of organelle genome-encoded 5S rRNAs including permuted molecules”. Nucleic Acids Res. 42 (22): 13764–13777. doi:10.1093/nar/gku1266. PMC 4267664. PMID 25429974.
- ^ Amunts, A; Brown, A; Bai, XC; Llácer, JL; Hussain, T; Emsley, P; Long, F; Murshudov, G et al. (March 2014). “Structure of the yeast mitochondrial large ribosomal subunit”. Science 343 (6178): 1485–1489. doi:10.1126/science.1249410. PMC 4046073. PMID 24675956.
- ^ Sharma, MR; Booth, TM; Simpson, L; Maslov, DA; Agrawal, RK (Jun 2009). “Structure of a mitochondrial ribosome with minimal RNA”. Proc Natl Acad Sci U S A 106 (24): 9637–9642. doi:10.1073/pnas.0901631106. PMC 2700991. PMID 19497863.
- ^ Brown, A; Amunts, A; Bai, XC; Sugimoto, Y; Edwards, PC; Murshudov, G; Scheres, SH; Ramakrishnan, V (Nov 2014). “Structure of the large ribosomal subunit from human mitochondria”. Science 346 (6210): 718–722. doi:10.1126/science.1258026. PMC 4246062. PMID 25278503.
- ^ Greber, BJ; Boehringer, D; Leibundgut, M; Bieri, P; Leitner, A; Schmitz, N; Aebersold, R; Ban, N (Nov 2014). “The complete structure of the large subunit of the mammalian mitochondrial ribosome”. Nature 515 (7526): 283–286. doi:10.1038/nature13895. hdl:20.500.11850/93239. PMID 25271403.